乳腺MRI影像组学模型对小乳腺癌诊断效能的研究
乳腺癌是女性最常见的恶性肿瘤和癌症死亡原因[1]。MRI是乳腺病变的重要检查手段,联合运用动态增强MRI(dynamic contrast-enhanced magnetic resonance imaging,DCE-MRI)和扩散加权成像(duffusion weighted imaging,DWI)诊断乳腺癌灵敏度和特异度高[2, 3]。临床上早期诊断T1期小乳腺癌(最大径≤20 mm)具有重要意义,不仅会影响治疗方案的制定,还能显著提高预后[4]。
T1N0M0期乳腺癌5年相对生存率高达97%-100%,而T4M0期仅为55%-71%[5]。随着影像组学的兴起,有研究发现基于乳腺MRI的影像组学模型可用于乳腺良恶性病灶的鉴别诊断,但是较小体积的病灶可能导致某些影像组学特征提取困难,影响分析结果[6, 7, 8, 9, 10, 11, 12]。
因此,本研究旨在探究基于DCE-MRI和DWI图像建立影像组学模型对小乳腺癌的诊断效能,并与放射科医师评估结果进行对比分析。
资料与方法
▶ 一般资料
回顾性分析2016年6月至2018年1月上海交通大学医学院附属仁济医院符合以下标准的女性患者。纳入标准:①经手术病理证实的乳腺小病灶(最大径≤20 mm);②术前行乳腺MRI检查;③手术和MRI检查间隔不超过4周。排除标准:①图像质量不佳,不能用于分析;②DWI图像上病灶显示不明显;③MRI检查前进行过穿刺活检、化学治疗、放射治疗或乳腺肿块切除术等治疗。
共入组192例患者,179例为单乳单发病灶,7例双侧乳房各1个病灶,6例为单侧乳房2个病灶,共205个病灶。205个病灶中恶性病灶86个,包括导管原位癌21个、浸润性导管癌47个、浸润性小叶癌12个、黏液癌5个、神经内分泌癌1个;良性病灶119个,包括纤维腺瘤46个、纤维囊性变24个、局灶性纤维化16个、腺病10个、乳头状瘤13个、非典型导管增生5个、其他(假血管瘤样间质增生、肌纤维母细胞瘤、脂肪坏死等)5个。
为更好地验证模型的泛化性能,本研究以入组时间为标准,将所有患者按照6∶4的比例分为训练集和测试集。训练集:入组时间2016年6月至2017年5月,共107例患者,116个病灶,年龄26~81,中位年龄56岁;测试集:入组时间2017年6月至2018年1月,共85例患者,89个病灶,年龄24-80,中位年龄55岁。
本研究经医院伦理委员会批准,所有患者均签署知情同意书(RA-2019-091)。
▶ MRI检查方法
采用荷兰Philips Achieva 3.0 T MRI扫描仪,4通道专用乳腺相控阵线圈。患者取俯卧位,双乳自然悬垂于线圈内。扫描序列和参数:①横轴面T1WI:重复时间(repetition time,TR)474 ms,回波时间(echo time,TE)8 ms,矩阵400×318,层厚3 mm;②横轴面T2WI:TR 4 700 ms,TE 120 ms,矩阵400×318,层厚3 mm;③矢状面T2WI脂肪抑制:采用精准频率反转恢复序列,TR 4 200 ms,TE 60 ms,矩阵308×244,层厚4 mm;④DWI:采用单次激发自旋回波平面回波成像序列,TR 4 500 ms,TE 85 ms,矩阵108×128,层厚3 mm,激励次数3,b=0、800 s/mm2;⑤横轴面DCE-MRI:采用T1高分辨率各向同性容积激发序列,TR 4.7 ms,TE 2.3 ms,矩阵320×340,层厚2 mm。对比剂使用钆喷替酸葡甲胺,剂量0.1 mmol/kg,注射流率2.0 ml/s,并以相同流率注入20 ml生理盐水冲管。对比剂注射前先扫描1个时相,对比剂注入结束后即刻开始连续扫描5个时相,每一时相间隔61 s。
▶ 影像组学分析
将所有患者的DCE-MRI和DWI图像传输至个人计算机,编写Matlab和Python 3.6代码分别进行图像预处理和影像组学分析。图像预处理包括:插值和配准。
影像组学分析包括:①病灶分割:运用半自动病灶分割法,由1名具有5年乳腺MRI诊断经验的放射科医师在DCE-MRI图像上利用长方体边界框包含所有层面上的病灶部分,与此同时,相对应的DWI图像上的病灶也被长方体边界框包含。迭代图割和k-均值聚类算法分别用于DCE-MRI和DWI图像上的病灶分割;②特征提取:分割后的病灶共提取413个影像学特征,包括58个形态学特征、340个纹理特征、7个血流动力学特征以及8个表观扩散系数(apparent diffusion coefficient,ADC)特征;③建立预测模型:基于scikit-learn 0.19.2.程序包(https://scikit-learn.org/0.19/install.html),运用梯度提升决策树(gradient boosting decision tree,GBDT)建立预测模型。
为了提升模型的泛化性能,基于训练后的模型,根据特征的重要性,采用机器学习中特征选择的方式进行降维。某一特征的重要性定义为该特征在单棵树上重要性的平均值,单棵树上的重要性表示为与该特征相关的节点分裂导致的均方误差下降之和。根据特征的重要性进行排序,并选择其中最重要的100个特征用于重新训练模型,并将重新训练所得GBDT模型用于测试集病灶的良恶性预测。
▶ 乳腺MRI放射医师分析
测试集患者乳腺MRI图像由1名具有16年乳腺MRI诊断经验的放射科医师在双盲的情况下,在后处理工作站(Ziostation2 ver.2.4.0.2;Ziosoft Inc.,Tokyo,Japan)上评估。
评估采用3种方法:①DCE-MRI评估:根据第5版美国放射学会MRI乳腺影像报告和数据系统(breast imaging reporting and data system,BI-RADS)分类标准[13],基于病灶形态学和时间信号强度曲线(time-signal intensity curve,TIC)类型,在DCE-MRI图像上对乳腺小病灶进行BI-RADS分类。测量病灶最大径时,选择病灶增强最明显图像并选择其最大层面,多发病灶则分别测量最大径。绘制TIC时,在病灶强化程度最高的区域放置感兴趣区(region of interest,ROI),多发病灶则分别绘制TIC。②DWI评估:参照T2WI和DCE-MRI图像,在DWI图像上选取病灶扩散受限最显著层面,在软件自动生成的相应ADC图中测量ADC值,由医师沿病灶边缘内侧手动绘制ROI,避开出血、坏死、囊变和脂肪组织,每个病灶测量3次,取最小值作为该病灶的ADC值。③DCE-MRI和DWI联合评估:参考学者Kul等[14]提出的诊断标准,在DCE-MRI图像上分别评估病灶在形态学和血流动力学两方面表现的良恶性,并记录DWI评估法中所得ADC值。
判断病灶良恶性:①DCE-MRI评估:定义BI-RADS 1、2、3类病变为良性,BI-RADS 4、5类病变为恶性;②DWI评估:根据病理学结果和ADC值,运用受试者操作特征(receiver operator characteristic,ROC)曲线得到最佳ADC阈值用于区分病灶良恶性;③DCE-MRI和DWI联合评估:基于上述所得ADC阈值,参考Kul等[14]提出的诊断标准,如果病灶在3项评估中(形态学、血流动力学、ADC值)至少有2项表现为恶性,该病灶则被认为是恶性,反之则为良性。
▶ 统计学分析
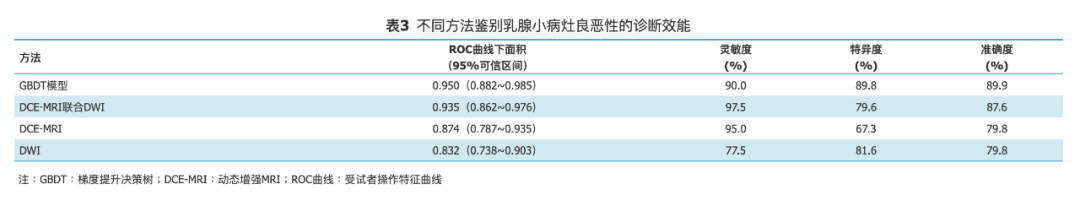
采用MedCalc 15.2.2软件进行统计分析。采用Kolmogorov-Smirnov对计量资料进行正态性检验,符合正态分布的计量资料用表示,不符合正态分布的计量资料用中位数(上、下四分位数)表示。计数资料以频数表示,采用卡方检验。以病理结果为金标准,运用ROC曲线分别计算影像组学和经验丰富的放射医师鉴别乳腺小病灶良恶性的灵敏度、特异度、准确度和ROC曲线下面积。
DeLong检验比较ROC曲线下面积,McNemar检验比较灵敏度、特异度和准确度。采用Kappa检验评估影像组学和DCE-MRI及DWI图像诊断小乳腺癌的一致性。P<0.05为差异有统计学意义。
结果
▶ 临床资料
205个病灶中有86个恶性病灶,其中训练集46个,测试集40个,2组相比差异无统计学意义(表1)。所有病灶最大径为12(3,20)mm。
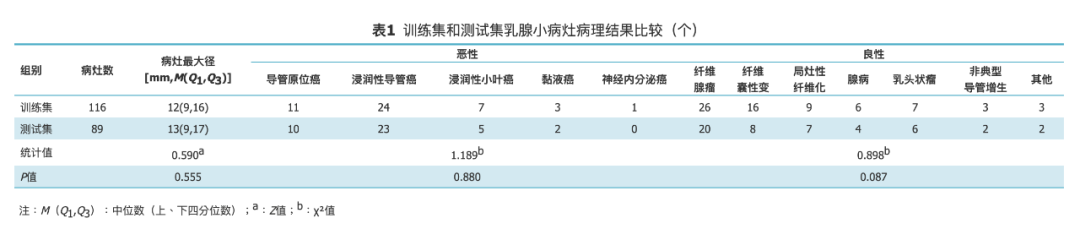
▶ 影像组学特征分析结果
根据所建GBDT模型特征的均方误差,本研究选取模型最重要的100个特征的前10位,其中包括3个ADC值特征、3个反映病灶边缘的纹理特征、2个血流动力学特征以及2个形态学特征(表2)。排名前5位中有2个ADC值特征、2个纹理特征、1个血流动力学特征。前10位特征中ADC值中位数均方误差最大。
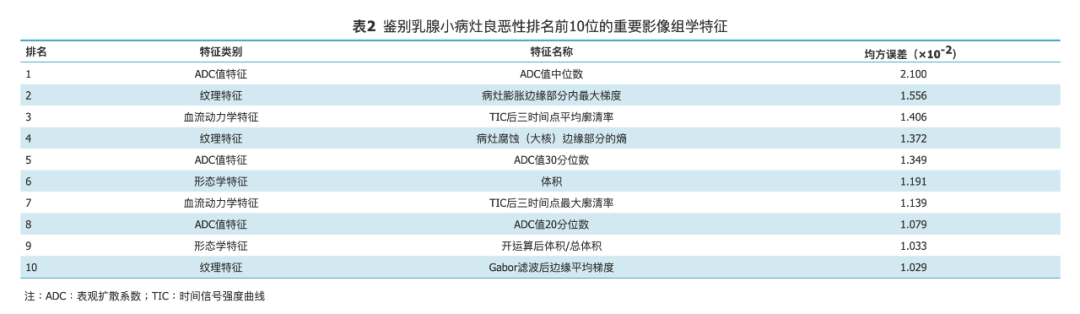
▶ 诊断效能评估
测试集中,放射科医师运用DWI方法鉴别乳腺病灶良恶性的ROC曲线,最佳ADC阈值为1.12×10-3 mm2/s。医师评估中,DCE-MRI联合DWI评估的ROC曲线下面积显著高于DCE-MRI、DWI孤立评估(Z=2.950,P=0.003;Z=2.491,P=0.013)。
DCE-MRI联合DWI的灵敏度显著高于DWI(χ²=6.125,P=0.008),而特异度显著高于DCE-MRI(χ²=4.167,P=0.031),准确度显著高于DCE-MRI(χ²=5.143,P=0.016),但与DWI差异无统计学意义(χ²=2.769,P= 0.092)。
GBDT影像组学模型分析的ROC曲线下面积与DCE-MRI联合DWI差异无统计学意义(Z=0.499,P=0.618),显著高于DCE-MRI和DWI孤立评估(Z=2.024,P=0.043;Z=2.772,P=0.006)(图1)。GBDT模型在最佳截断点灵敏度、特异度和准确度与DCE-MRI联合DWI差异均无统计学意义(χ²=0.800、2.286和0.083,P均>0.05),而特异度显著高于DCE-MRI(χ²=7.692,P=0.003),准确度显著高于DWI(χ²=4.267,P=0.035)(表3)。
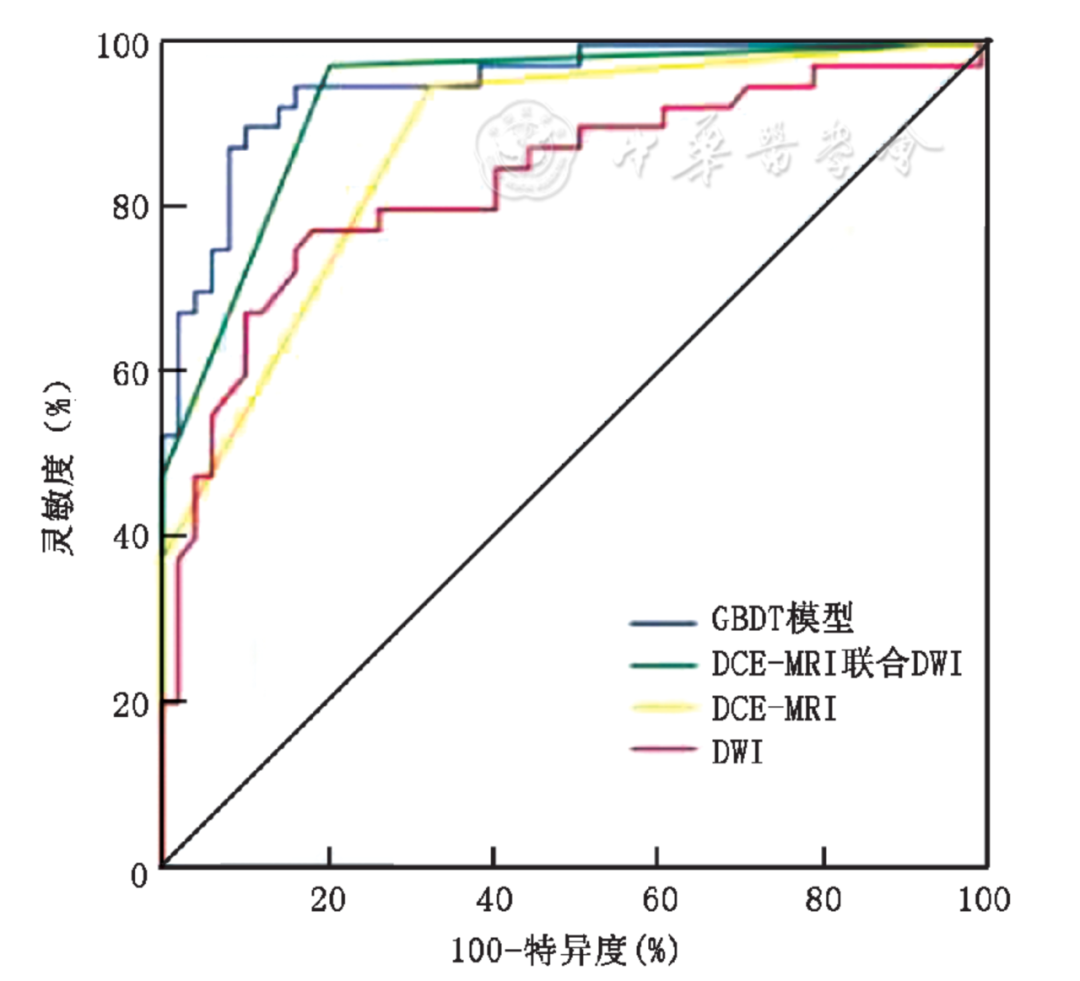
图1.影像组学梯度提升决策树(GBDT)模型、动态增强MRI(DCE-MRI)联合扩散加权成像(DWI)、DCE-MRI以及DWI鉴别乳腺小病灶良恶性的受试者操作特征(ROC)曲线,曲线下面积分别为0.950、0.935、0.874、0.832
Kappa检验显示GBDT模型和放射科医师联合DCE-MRI及DWI图像诊断小乳腺癌的一致性较高,Kappa值为0.754,95%可信区间为0.620~0.889。图2, 3, 4, 5和图6, 7, 8, 9分别显示了GBDT影像组学模型准确预测乳腺恶性和良性小病灶病例。
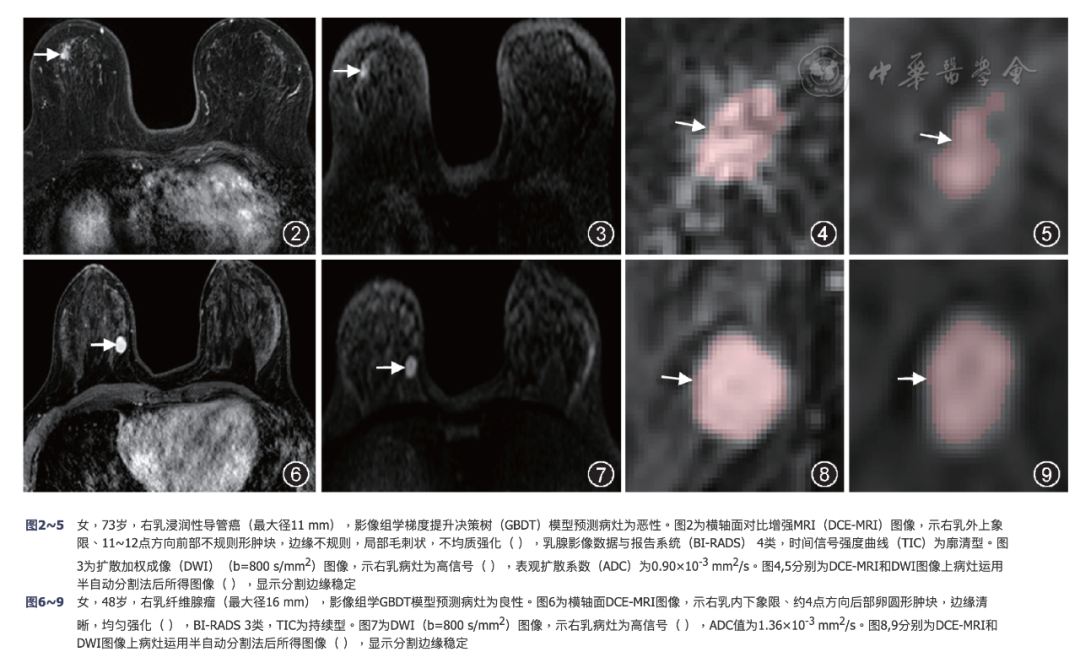
图2-5
女,73,右乳浸润性导管癌(最大径11 mm),影像组学梯度提升决策树(GBDT)模型预测病灶为恶性。图2为横轴面对比增强MRI(DCE-MRI)图像,示右乳外上象限、11-12点方向前部不规则形肿块,边缘不规则,局部毛刺状,不均质强化( ),乳腺影像数据与报告系统(BI-RADS)4类,时间信号强度曲线(TIC)为廓清型。图3为扩散加权成像(DWI)(b=800 s/mm2)图像,示右乳病灶为高信号( ),表观扩散系数(ADC)为0.90×10-3 mm2/s。图4,5分别为DCE-MRI和DWI图像上病灶运用半自动分割法后所得图像( ),显示分割边缘稳定
图6-9
女,48,右乳纤维腺瘤(最大径16 mm),影像组学GBDT模型预测病灶为良性。图6为横轴面DCE-MRI图像,示右乳内下象限、约4点方向后部卵圆形肿块,边缘清晰,均匀强化( ),BI-RADS 3类,TIC为持续型。图7为DWI(b=800 s/mm2)图像,示右乳病灶为高信号( ),ADC值为1.36×10-3 mm2/s。图8,9分别为DCE-MRI和DWI图像上病灶运用半自动分割法后所得图像( ),显示分割边缘稳定
讨论
既往研究显示,运用MRI图像基于计算机辅助诊断技术(如纹理分析)鉴别乳腺较小病灶良恶性的ROC曲线下面积为0.76-0.82[15, 16]。本研究结果显示,基于DCE-MRI和DWI图像建立的GBDT影像组学模型对于诊断最大径≤20 mm的乳腺癌具有很好的诊断效能,ROC曲线下面积为0.950,灵敏度、特异度和准确度分别为90.0%、89.8%和89.9%,与联合DCE-MRI和DWI图像评估的诊断效能相比差异无统计学意义(P>0.05),且优于单独采用DCE-MRI或DWI图像的诊断效能。既往研究显示,联合DCE-MRI和DWI图像诊断乳腺病灶良恶性的效能要高于单独采用DCE-MRI或DWI图像[2,14,17]。
本研究显示,对于诊断小乳腺癌,在放射科医师的3种评估法中,DCE-MRI联合DWI方法的ROC曲线下面积、灵敏度、特异度和准确度最大,与Pinker等[2]对110个较大乳腺病灶(6-100 mm)的研究结果一致。Bickelhaupt等[10]研究显示,基于MRI平扫和DWI图像的影像组学分析在鉴别乳腺病灶良恶性方面表现良好,但诊断效能低于经验丰富的放射科医师,另一项基于MRI扩散峰度成像的研究显示,影像组学模型能可靠地鉴别乳腺病灶的良恶性,ROC曲线下面积为0.911[9]。与本研究不同的是,上述2项影像组学研究中并没有使用DCE-MRI图像,所建模型可能会丢失一些重要的影像组学特征。
本研究显示,前10位最重要影像组学特征中,ADC值特征、反映病灶边缘的纹理特征、血流动力学特征以及形态学特征均起到了重要作用,其中尤以前3者为甚,而ADC值中位数为最重要的影像组学特征。上述特征也是放射科医师分析乳腺MRI图像的主要关注点和诊断依据。这可能也是本研究中基于DCE-MRI和DWI图像的影像组学模型与放射科医师的诊断效能相似,并且具有较高的一致性的原因。这也进一步说明,本研究建立的影像组学模型获取了放射科医师进行乳腺MRI图像分析时使用的大部分关键影像特征信息。
Chen等[18]研究发现,运用全容积ADC直方图鉴别乳腺病灶良恶性时,ADC值中位数得到最高的ROC曲线下面积。Suo等[19]运用全容积ADC直方图研究乳腺癌异质性时发现,最小ADC值和第25百分位数ADC值比ADC值中位数或平均值能更准确鉴别乳腺病灶的良恶性。本研究显示ADC值中位数为最重要的影像组学特征。这些差异可能是由于不同研究所用病灶提取方式不同导致,但总体来看,较低百分位数ADC值相对于较高百分位数ADC值鉴别良恶性肿瘤更有价值,这可能是因为较低百分位数ADC值代表病灶中水分子扩散受限更明显、肿瘤细胞更密集区域。
血流动力学特征是乳腺病灶良恶性鉴别的重要依据[20, 21],这与本研究结果一致。Schnall等[20]研究发现,乳腺廓清型TIC肿块为恶性肿瘤的可能性是平台型的5倍。有多项研究证实,乳腺MRI图像中肿块的边缘特征是预测病灶良恶性孤立的、最重要的特征[22, 23]。
本研究所建影像组学模型前10位最重要特征中反映病灶边缘的纹理特征占3项,其中两项居前5位,显示了反映病灶边缘的纹理特征对于鉴别病灶良恶性的重要性。虽然早有研究表明,纹理特征对于乳腺病灶良恶性鉴别具有重要意义[24, 25, 26],但本研究中反映病灶全局的纹理特征并没有出现在前10位最重要影像组学特征中,笔者分析可能的原因有两个方面,首先本研究的影像组学模型全面综合分析了大量影像学特征,反映病灶全局的纹理特征重要性相对较低;其次本研究对象为最大径≤20 mm的小病灶,可提取特征的体素较少,从而导致反映病灶全局的纹理特征提取困难,且有一定的误差,可能影响最终的分析结果[9]。
本研究还存在一些局限性:①样本量偏小,增加样本量可能会进一步提高模型的诊断效能;②病灶分割采用半自动法,操作者经验可能影响结果,探索病灶全自动分割是今后的研究方向;③训练集和测试集的MRI图像采集使用同1台MRI扫描仪,今后需要运用不同设备采集的图像,甚至多中心研究进一步验证模型的效能。
综上所述,基于乳腺DCE-MRI和DWI图像建立的影像组学模型对于小乳腺癌具有较高的诊断效能,其与放射科医师联合DCE-MRI和DWI图像评估的诊断效能相似,且优于单独采用DCE-MRI或DWI图像评估的结果。
参考文献
1.BrayF, FerlayJ, SoerjomataramI, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(06):394-424. DOI: 10.3322/caac.21492.
2.PinkerK, MoyL, SuttonEJ, et al. Diffusion-weighted imaging with apparent diffusion coefficient mapping for breast cancer detection as a stand-alone parameter: comparison with dynamic contrast-enhanced and multiparametric magnetic resonance imaging[J]. Invest Radiol, 2018, 53(10):587-595. DOI: 10.1097/RLI.0000000000000465.
3.中华医学会影像技术分会,中华医学会放射学分会.乳腺影像检查技术专家共识[J].中华放射学杂志,2016,50(08):561-565.DOI: 10.3760/cma.j.issn.1005-1201.2016.08.001.
4.NatarajanB, SpiegelD, NicholsEM, et al. Findings on surveillance imaging after preoperative partial breast irradiation for early stage breast cancer[J]. Int J Radiat Oncol Biol Phys, 2018, 102(04):1374-1381. DOI: 10.1016/j.ijrobp.2018.05.059.
5.SantM, AllemaniC, BerrinoF, et al. Breast carcinoma survival in Europe and the United States[J]. Cancer, 2004, 100(04):715-722. DOI: 10.1002/cncr.20038.
6.LambinP, LeijenaarRTH, DeistTM, et al. Radiomics: the bridge between medical imaging and personalized medicine[J]. Nat Rev Clin Oncol, 2017, 14(12):749-762. DOI: 10.1038/nrclinonc.2017.141.
7.史张,李晶,边云,等.影像组学在临床精确诊疗中的研究进展[J].中华放射学杂志,2018,52(10):801-804.DOI: 10.3760/cma.j.issn.1005-1201.2018.10.019.
8.马文娟,赵玉梅,季宇,等.基于乳腺X线影像组学特征的预测模型在鉴别三阴型与非三阴型乳腺癌中的价值[J].中华放射学杂志,2018,52(11):842-846.DOI: 10.3760/cma.j.issn.1005-1201.2018.11.006.
9.BickelhauptS, JaegerPF, LaunFB, et al. Radiomics based on adapted diffusion kurtosis imaging helps to clarify most mammographic findings suspicious for cancer[J]. Radiology, 2018, 287(03):761-770. DOI: 10.1148/radiol.2017170273.
10.BickelhauptS, PaechD, KickingerederP, et al. Prediction of malignancy by a radiomic signature from contrast agent-free diffusion MRI in suspicious breast lesions found on screening mammography[J]. J Magn Reson Imaging, 2017, 46(02):604-616. DOI: 10.1002/jmri.25606.
11.ValdoraF, HoussamiN, RossiF, et al. Rapid review: radiomics and breast cancer[J]. Breast Cancer Res Treat, 2018, 169(02):217-229. DOI: 10.1007/s10549-018-4675-4.
12.胡斌,徐克,张立娜,等.基于表观扩散系数图像的影像组学模型对MRI乳腺影像报告与数据系统4类病变良恶性的鉴别诊断价值[J].中华放射学杂志,2017,51(12):922-925.DOI: 10.3760/cma.j.issn.1005-1201.2017.12.007.
13.RaoAA, FeneisJ, LalondeC, et al. A pictorial review of changes in the BI-RADS fifth edition[J]. Radiographics, 2016, 36(03):623-639. DOI: 10.1148/rg.2016150178.
14.KulS, CansuA, AlhanE, et al. Contribution of diffusion-weighted imaging to dynamic contrast-enhanced MRI in the characterization of breast tumors[J]. AJR Am J Roentgenol, 2011, 196(01):210-217. DOI: 10.2214/AJR.10.4258.
15.SchlossbauerT, LeinsingerG, WismullerA, et al. Classification of small contrast enhancing breast lesions in dynamic magnetic resonance imaging using a combination of morphological criteria and dynamic analysis based on unsupervised vector-quantization[J]. Invest Radiol, 2008, 43(01):56-64. DOI: 10.1097/RLI.0b013e3181559932.
16.NagarajanMB, HuberMB, SchlossbauerT, et al. Classification of small lesions in breast MRI: evaluating the role of dynamically extracted texture features through feature selection[J]. J Med Biol Eng, 2013, 33(1) 10.5405/jmbe.1183. DOI: 10.5405/jmbe.1183.
17.ZhangL, TangM, MinZ, et al. Accuracy of combined dynamic contrast-enhanced magnetic resonance imaging and diffusion-weighted imaging for breast cancer detection: a meta-analysis[J]. Acta Radiol, 2016, 57(06):651-660. DOI: 10.1177/0284185115597265.
18.ChenY, WuB, LiuH, et al. Feasibility study of dual parametric 2D histogram analysis of breast lesions with dynamic contrast-enhanced and diffusion-weighted MRI[J]. J Transl Med, 2018, 16(01):325. DOI: 10.1186/s12967-018-1698-x.
19.SuoS, ZhangK, CaoM, et al. Characterization of breast masses as benign or malignant at 3.0 T MRI with whole-lesion histogram analysis of the apparent diffusion coefficient[J]. J Magn Reson Imaging, 2016, 43(04):894-902. DOI: 10.1002/jmri.25043.
20.SchnallMD, BlumeJ, BluemkeDA, et al. Diagnostic architectural and dynamic features at breast MR imaging: multicenter study[J]. Radiology, 2006, 238(01):42-53. DOI: 10.1148/radiol.2381042117.
21.El KhouliRH, MacuraKJ, KamelIR, et al. 3-T dynamic contrast-enhanced MRI of the breast: pharmacokinetic parameters versus conventional kinetic curve analysis[J]. AJR Am J Roentgenol, 2011, 197(06):1498-1505. DOI: 10.2214/AJR.10.4665.
22.KawaiM, KataokaM, KanaoS, et al. The value of lesion size as an adjunct to the BI-RADS-MRI 2013 descriptors in the diagnosis of solitary breast masses[J]. Magn Reson Med Sci, 2018, 17(03):203-210. DOI: 10.2463/mrms.mp.2017-0024.
23.SzaboBK, AspelinP, WibergMK, et al. Dynamic MR imaging of the breast. analysis of kinetic and morphologic diagnostic criteria[J]. Acta Radiol, 2003, 44(04):379-386. DOI: 10.1034/j.1600-0455.2003.00084.x.
24.GibbsP, TurnbullLW. Textural analysis of contrast-enhanced MR images of the breast[J]. Magn Reson Med, 2003, 50(01):92-98. DOI: 10.1002/mrm.10496.
25.NieK, ChenJH, YuHJ, et al. Quantitative analysis of lesion morphology and texture features for diagnostic prediction in breast MRI[J]. Acad Radiol, 2008, 15(12):1513-1525. DOI: 10.1016/j.acra.2008.06.005.
26.ChenW, GigerML, LiH, et al. Volumetric texture analysis of breast lesions on contrast-enhanced magnetic resonance images[J]. Magn Reson Med, 2007, 58(03):562-571. DOI: 10.1002/mrm.21347.